Relatório - Prova Prática 2
Análise de Séries Temporais
Allan Vieira 14/0128492
Junho de 2018
1) Análise da temperatura global em dados registrados pela NASA (Agência Espacial Norte-Americana) e pela NOAA (Administração Atmosférica e Oceânica Nacional), desde 1980.
Contextualização do problema:
Se \(W_t\) representa a temperatura global observada no mês \(t\) (média mensal, em graus Celsius) e \(\mu\) denota uma temepratura média global constante, o arquivo \(\verb|TempGlobal.csv|\) encontrado no Aprender contém as séries históricas dos desvios \(X_t = Y_t - \mu\), registradas pela NASA (Agência Espacial Norte-Americana) e pela NOAA (Administração Atmosférica e Oceânica Nacional), desde 1980. Para a NASA, o período base para a estomativa da média \(\mu\) compreende o período de 1951 a 1980. Para a NOAA, o período base refere-se ao séc. XX.
item i) Identificar um processo ARIMA para essas séries temporais, justificando a escolha por meio de critérios técnicos.
Dados brutos antes do tratamento:
Após removermos a coluna de índices dos dados diretamente no Excel, temos:
# ----- leitura e carregamento
library(tibbletime)
library(dplyr)
library(ggplot2)
library(readr)
library(lubridate)
library(reshape2)
temp.loc <- "/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/TempGlobal_alt.csv"
temp <- read_csv(temp.loc)
# ----- data preparation
# eliminando os NA's
# e formatando Data para tibbletime
temp2 <- temp %>%
na.omit() %>%
dplyr::mutate(Date = lubridate::ymd(Date)) %>% # usando ymd do lubridate
as_tbl_time(index = Date)
# head(matter3)
temp2Nosso interesse é obter o melhor modelo em termos técnicos, ou seja, obter um modelo ARIMA com o melhor desempenho de predição. Por isso, nossa estratégia não será a de testar a estacionariedade da série, mas sim ajustar modelos com a série sem diferenciar e com a série diferenciada e verificar seus resultados em termos do Mean Absolute Percentage Error (MAPE) na predição de um passo.
Inicialmente, procedemos a uma análise de ordem exploratória nos dados para termos uma ideia do comportamento da série.
Começamos pela plotagem das séries.
Gráficos das séries mensais:
# crinado as ts
X.NOAA_ts <- ts(temp2 %>% dplyr::select(X.NOAA), start = c(1880,1), end =c(2016,12), frequency = 12)
# o fato de ser todo dia 15 nessas series nao importa -- a frequencia indica que sao 12 medicoes ao ano
X.NASA_ts <- ts(temp2 %>% dplyr::select(X.NASA), start = c(1880,1), end = c(2016,12), frequency = 12)library(forecast)
library(highcharter)
library(ggplot2)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
temp2_ts <- cbind(X.NOAA_ts, X.NASA_ts)
hchart(temp2_ts) %>%
hc_colors(plotly_palette[1:2]) %>%
hc_title(text = "Série Histórica Mensal dos Desvios de Temperatura (1880 - 2016)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))Faremos também o plot das séries anuais conforme segue.
Gráficos das séries anuais:
# ----- plots serie:
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728')
# --- serie:
# highcharts:
temp_ano <- temp2 %>%
as_period("1 y") # haverah uma linha para cada ano
# crinado as ts
X.NOAA_ano_ts <- ts(temp_ano %>% dplyr::select(X.NOAA), start = 1880, end = 2016, frequency = 1)
X.NASA_ano_ts <- ts(temp_ano %>% dplyr::select(X.NASA), start = 1880, end = 2016, frequency = 1)
temp_ano_ts <- cbind(X.NOAA_ano_ts, X.NASA_ano_ts)
hchart(temp_ano_ts) %>%
hc_colors(plotly_palette[1:2]) %>%
hc_title(text = "Série Histórica Anual dos Desvios de Temperatura (1880 - 2016)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))Finalizando nossa análise exploratória, fazemos os plots da Função de Autocorrelação (FAC) e da Função de Autocorrelação Parcial (FACP).
Plots da FAC e FACP:
# funcoes para plotar ACF e PACF nos moldes do ggplot2
# adaptado de: https://stackoverflow.com/questions/42753017/adding-confidence-intervals-to-plotted-acf-in-ggplot2
gg_acf <- function(ts, title="FAC", lag=1, lag.max=50){
ts_acf <- acf(ts, lag = lag, lag.max = lag.max, na.action = na.pass, plot=FALSE)
conf_lims <- c(-1,1)*qnorm(0.975)/sqrt(ts_acf$n.used)
ts_acf$acf %>%
# tibble::as_tibble() %>% dplyr::mutate(lags = 0:(n()-1)) %>% # acf começa em zero e termina em n-1
tibble::as_tibble() %>% dplyr::mutate(lags = 0:(lag.max)) %>% # acf começa em zero e termina em lag.max
ggplot2::ggplot(aes(x=lags, y = V1)) + ggplot2::scale_x_continuous(breaks=seq(0,lag.max,lag.max/10)) +
ggplot2::geom_hline(yintercept=conf_lims, lty=2, col='red', size=0.3) +
ggplot2::labs(y="Autocorrelations", x="Lag", title=title) +
ggplot2::geom_segment(aes(xend=lags, yend=0)) + theme_minimal()
#+ ggplot2::geom_point()
}
gg_pacf <- function(ts, title="FACP", lag=1, lag.max=50){
# ts = temp[,c("Date", "X.NASA")]
ts_pacf <- pacf(ts, lag = lag, lag.max = lag.max, na.action = na.pass, plot=FALSE)
# corrigindo no zero:
ts_pacf$acf <- c("0"=1, ts_pacf$acf)
conf_lims <- c(-1,1)*qnorm(0.975)/sqrt(ts_pacf$n.used)
ts_pacf$acf %>% # tb eh acf o objeto dentro do output de pacf
# tibble::as_tibble() %>% dplyr::mutate(lags = 1:n()) %>% # pacf começa em 1 e termina em n
tibble::as_tibble() %>% magrittr::set_colnames(c("V1")) %>% # para funcionar apos a inclusao do zero
dplyr::mutate(lags = 0:(lag.max)) %>% # acf começa em zero e termina em lag.max
ggplot2::ggplot(aes(x=lags, y = V1)) + ggplot2::scale_x_continuous(breaks=seq(0,lag.max,lag.max/10)) +
ggplot2::geom_hline(yintercept=conf_lims, lty=2, col='red', size=0.3) +
ggplot2::labs(y="Partial Autocorrelations", x="Lag", title=title) +
ggplot2::geom_segment(aes(xend=lags, yend=0)) + theme_minimal()
#+ ggplot2::geom_point()
}library(gridExtra)
grid.arrange(gg_acf(temp[,c("X.NOAA")], title = "FAC - NOAA", lag.max=12*5), gg_pacf(temp[,c("X.NOAA")], title = "FACP - NOAA", lag.max=12*5), ncol=2)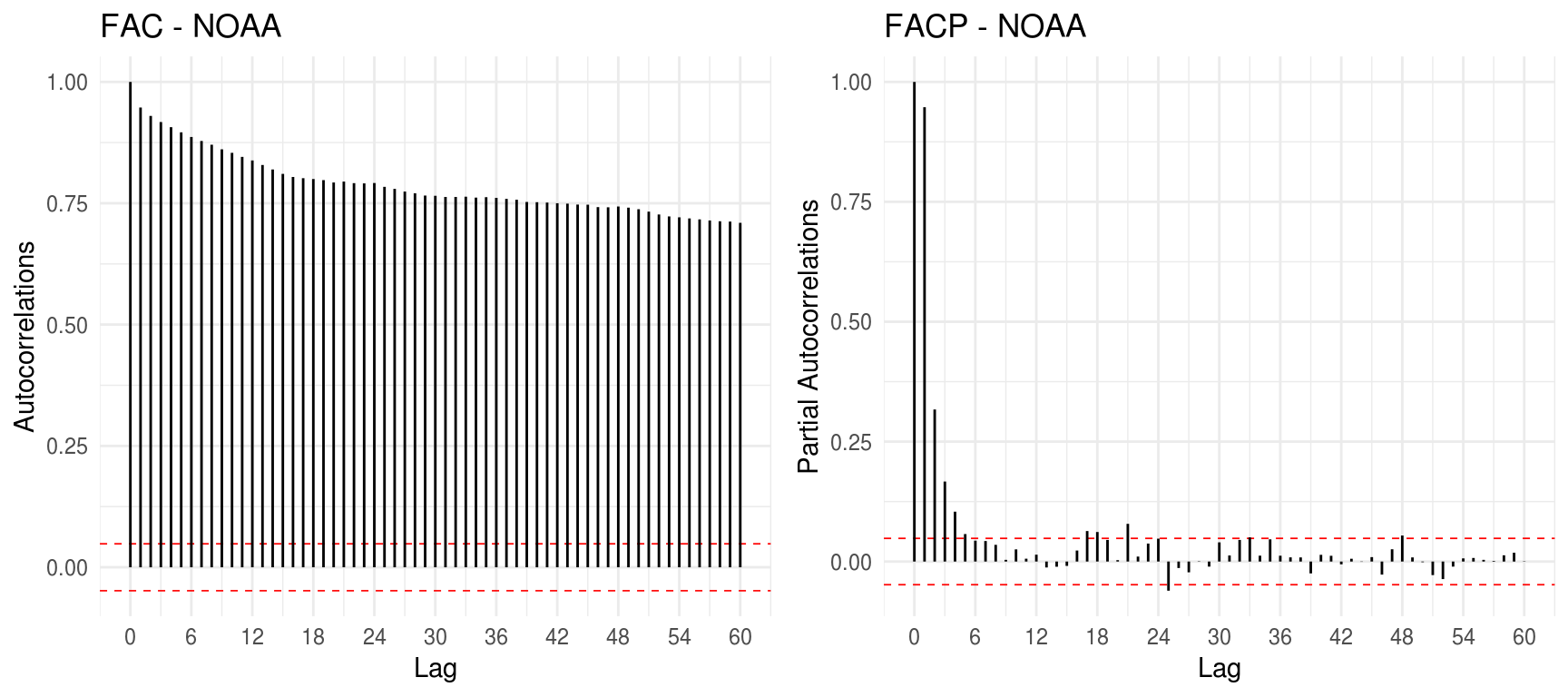
library(gridExtra)
grid.arrange(gg_acf(temp[,c("X.NASA")], title = "FAC - NASA", lag.max=12*5), gg_pacf(temp[,c("X.NASA")], title = "FACP - NASA", lag.max=12*5), ncol=2)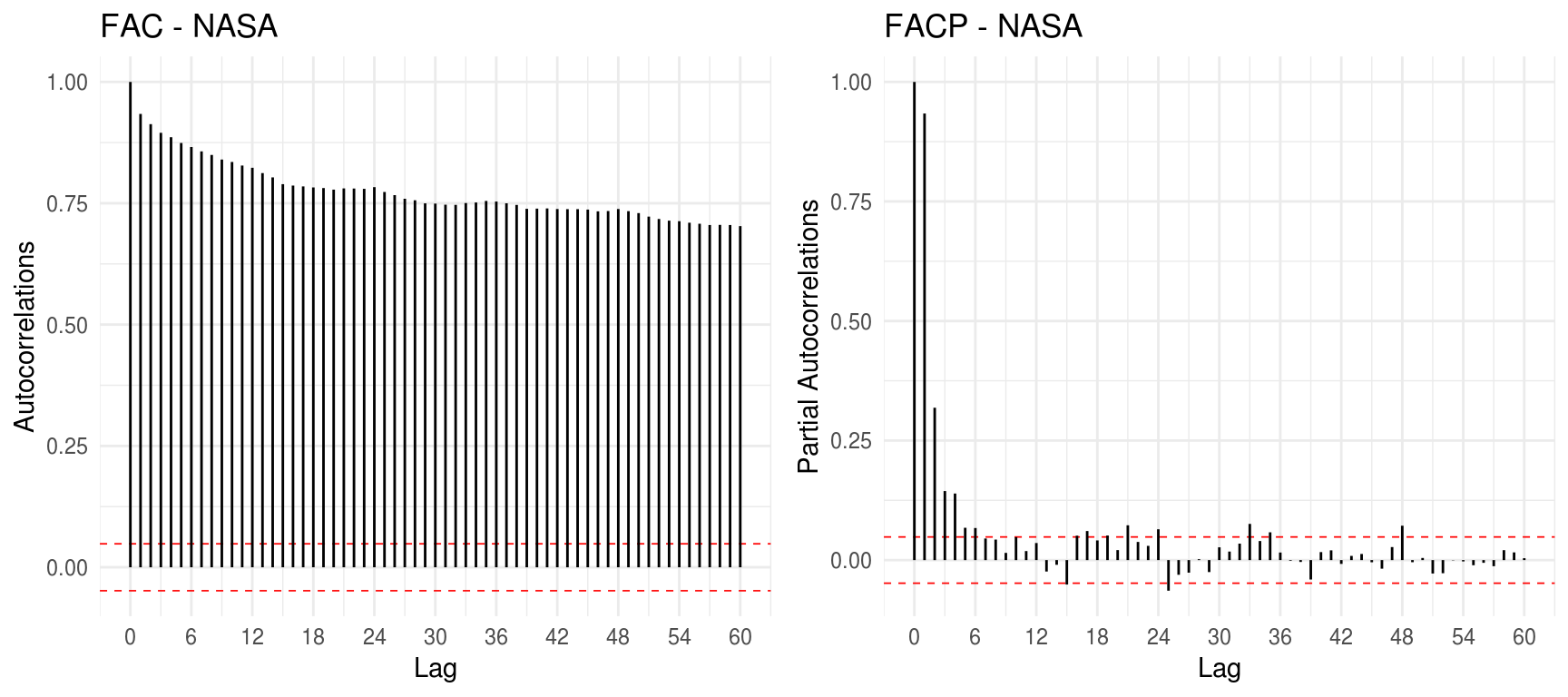
Fica claro que ambas as séries não aparentam ter estacionariedade, o que é reforçado por uma possível tendência nos gráficos mensais e anuais. Por outro lado, não há evidências de sazonalidade, indicando a adequabilidade de um modelo ARIMA não-sazonal para o caso. Como nosso objetivo é obter o modelo com base na abordagem empírica de performance na predição, deixaremos que eventuais restrições relacionadas a não estacionariedade reflitam nos resultados ao selecionarmos os melhores modelos. Utilizaremos o Mean Absolute Percentage Error (MAPE) de um passo a frente como critério de otimalidade, tomando como base os últimos 12 meses, para confeccionar um grid que nos apontará os modelos candidatos.
Modelos sem diferenciação [ARIMA(p,0,q)]
# -----------------------------------------------------------------------
# Identificao da ordem do processo
# com base no erro de previsao 1 passo a frente
# relativo as ultimas 12 observacoes
# -----------------------------------------------------------------------
X.0 <- temp[,c("X.NOAA"), drop=TRUE] # extrair somente os valores como vetor mesmo
N <- nrow(temp[,c("X.NOAA")])
X.0 <- temp[,c("X.NASA"), drop=TRUE]
N <- nrow(temp[,c("X.NASA")])
p.max = 7
q.max = 3
MAPE <- matrix(0, p.max+1, q.max+1)
for (p in 0:p.max){
for (q in 0:q.max){
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
# fit <- arima(X, order=c(p, 1, q),xreg=1:n) #Certo
fit <- arima(X, order=c(p, 0, q),xreg=1:n) #Certo
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
MAPE[(p+1),(q+1)] <- 100*(mean(abs((previsto-observado)/observado)))
}
}
MAPE_nodiff_NOAA <- MAPE
MAPE_nodiff_NASA <- MAPE
readr::write_tsv(as.data.frame(MAPE_nodiff_NOAA), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_NOAA.txt")
readr::write_tsv(as.data.frame(MAPE_nodiff_NASA), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_NASA.txt")Temos os seguintes grids resultantes do algoritmo de otimização:
MAPE_nodiff_NOAA <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_NOAA.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(., 4) %>%
`rownames<-`(paste0("p=", 0:7))
MAPE_nodiff_NASA <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_NASA.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(.,4) %>%
`rownames<-`(paste0("p=", 0:7))Para cada série analisada, elencamos os 3 melhores modelo, os quais são destacados nos respectivos grids.
library(kableExtra)
library(knitr)
MAPE_nodiff_NOAA %>%
dplyr::mutate(
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` == 7.9136, plotly_palette[3],
ifelse(`q=1` < 8.03 & `q=1` != 7.9136, plotly_palette[1], "white")),
color = dplyr::if_else(`q=1` < 8.03, "white", "black")),
# `q=0` = cell_spec(`q=0`,
# background = ifelse(`q=0` < 8.03, plotly_palette[1], "white"),
# color = dplyr::if_else(`q=0` < 8.03, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid NOAA") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 44.3026 | 26.5005 | 19.4825 | 15.9024 |
| p=1 | 8.8955 | 8.3984 | 8.3997 | 8.4257 |
| p=2 | 8.0871 | 8.4045 | 8.0565 | 8.0970 |
| p=3 | 8.0230 | 8.4283 | 8.4320 | 8.2356 |
| p=4 | 8.2474 | 7.9136 | 8.2910 | 8.2295 |
| p=5 | 8.0991 | 8.0262 | 8.1548 | 8.5262 |
| p=6 | 8.2188 | 8.0145 | 8.0457 | 8.1854 |
| p=7 | 8.5408 | 8.1308 | 8.1822 | 8.2348 |
MAPE_nodiff_NASA %>%
dplyr::mutate(
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` == 11.2289, plotly_palette[3],
ifelse(`q=1` < 11.3 & `q=1` != 11.2289, plotly_palette[1], "white")),
color = dplyr::if_else(`q=1` < 8.03, "white", "black")),
`q=2` = cell_spec(`q=2` ,
background = ifelse(`q=2` < 11.5, plotly_palette[2], "white"),
color = dplyr::if_else(`q=2` < 11.5, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black"),
`q=0` = cell_spec(`q=0` ,
background = ifelse(`q=0` < 11.3, plotly_palette[2], "white"),
color = dplyr::if_else(`q=0` < 11.3, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid NASA") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 47.3755 | 29.3741 | 21.3981 | 18.0524 |
| p=1 | 11.2414 | 12.2492 | 12.3714 | 12.1266 |
| p=2 | 11.8039 | 12.1782 | 12.13 | 11.6927 |
| p=3 | 11.8599 | 11.5415 | 12.1346 | 11.6237 |
| p=4 | 12.3878 | 11.2289 | 11.6482 | 11.7429 |
| p=5 | 12.5041 | 12.0852 | 11.4171 | 11.5059 |
| p=6 | 12.4723 | 11.9215 | 11.7652 | 11.4321 |
| p=7 | 12.4225 | 11.781 | 11.9779 | 11.6692 |
Após selecionarmos os melhores modelos no caso da série não diferenciada, passamos para a confecção dos grids de desempenho para a série diferenciada.
Modelos com diferenciação de ordem 1 [ARIMA(p,1,q)]
Novamente, utilizaremos o Mean Absolute Percentage Error (MAPE) de um passo a frente como critério de otimalidade e tomando como base os últimos 12 meses para confeccionar um grid que nos apontará os modelos candidatos.
# -----------------------------------------------------------------------
# Identificao da ordem do processo
# com base no erro de previsao 1 passo a frente
# relativo as ultimas 12 observacoes
# -----------------------------------------------------------------------
X.0 <- temp[,c("X.NOAA"), drop=TRUE] # extrair somente os valores como vetor mesmo
N <- nrow(temp[,c("X.NOAA")])
X.0 <- temp[,c("X.NASA"), drop=TRUE]
N <- nrow(temp[,c("X.NASA")])
p.max = 7
q.max = 3
MAPE <- matrix(0, p.max+1, q.max+1)
for (p in 0:p.max){
for (q in 0:q.max){
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
fit <- arima(X, order=c(p, 1, q),xreg=1:n) #Certo
# fit <- arima(X, order=c(p, 0, q),xreg=1:n) #Certo
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
MAPE[(p+1),(q+1)] <- 100*(mean(abs((previsto-observado)/observado)))
}
}
MAPE_diff_NOAA <- MAPE
MAPE_diff_NASA <- MAPE
readr::write_tsv(as.data.frame(MAPE_diff_NOAA), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_NOAA.txt")
readr::write_tsv(as.data.frame(MAPE_diff_NASA), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_NASA.txt")Temos os seguintes grids resultantes do algoritmo de otimização:
MAPE_diff_NOAA <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_NOAA.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(., 4) %>%
`rownames<-`(paste0("p=", 0:7))
MAPE_diff_NASA <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_NASA.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(.,4) %>%
`rownames<-`(paste0("p=", 0:7))Para cada série analisada, elencamos os 3 melhores modelo, os quais são destacados nos respectivos grids.
library(kableExtra)
library(knitr)
MAPE_diff_NOAA %>%
dplyr::mutate(
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` == 8.0038, plotly_palette[3],
ifelse(`q=1` < 8.07 & `q=1` != 7.9136, plotly_palette[1], "white")),
color = dplyr::if_else(`q=1` < 8.03, "white", "black")),
# `q=0` = cell_spec(`q=0`,
# background = ifelse(`q=0` < 8.03, plotly_palette[1], "white"),
# color = dplyr::if_else(`q=0` < 8.03, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid NOAA") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 8.1550 | 9.7389 | 10.0644 | 10.1234 |
| p=1 | 8.4431 | 10.1098 | 8.1507 | 8.1945 |
| p=2 | 9.0048 | 8.0659 | 8.2027 | 8.1980 |
| p=3 | 9.1085 | 8.0038 | 8.2143 | 8.1881 |
| p=4 | 9.4822 | 8.0633 | 8.1947 | 8.2052 |
| p=5 | 9.7837 | 8.0869 | 8.0813 | 8.1992 |
| p=6 | 10.0689 | 8.1458 | 8.1721 | 8.2124 |
| p=7 | 10.1352 | 8.2839 | 8.1169 | 8.1729 |
MAPE_diff_NASA %>%
dplyr::mutate(
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` == 11.1388, plotly_palette[3],
ifelse(`q=1` < 11.3 & `q=1` != 11.2289, plotly_palette[2], "white")),
color = dplyr::if_else(`q=1` < 8.03, "white", "black")),
`q=2` = cell_spec(`q=2` ,
background = ifelse(`q=2` == 11.1388, plotly_palette[3], "white"),
color = dplyr::if_else(`q=2` < 11.3, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black"),
`q=0` = cell_spec(`q=0` ,
background = ifelse(`q=0` < 11.3, plotly_palette[2], "white"),
color = dplyr::if_else(`q=0` < 11.3, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid NASA") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 11.5822 | 13.6448 | 13.4456 | 13.2608 |
| p=1 | 12.1087 | 12.9305 | 11.5798 | 11.7431 |
| p=2 | 12.2334 | 11.2099 | 11.7838 | 11.5475 |
| p=3 | 13.4908 | 11.197 | 11.1388 | 11.8399 |
| p=4 | 13.5378 | 11.8974 | 11.9816 | 11.7654 |
| p=5 | 13.575 | 11.9675 | 11.8125 | 11.9070 |
| p=6 | 13.7499 | 11.9912 | 11.9909 | 12.0445 |
| p=7 | 13.627 | 11.995 | 11.9424 | 12.0092 |
No caso da série NOAA os melhores modelos apareceram no caso sem diferenciar, ao passo que na série NASA, os melhores modelos estão na série com diferenciação de 1ª ordem. Portanto, utilizando como critério o desempenho medido pelo MAPE de um passo, no caso NOAA, o modelo escolhido é um ARIMA(4,0,1) (ou ARMA(4,1)). No caso da série NASA, o modelo escolhido é um ARIMA(3,1,2).
Plot valores preditos vs. observados nos modelos selecionados:
Abaixo apresentamos os plots comparando os valores observados e valores preditos para as últimas 12 observações para os modelos selecionados.
X.0 <- X.NOAA_ts
N <- length(X.0)
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
fit <- arima(X, order=c(4, 0, 1),xreg=1:n)
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
hchart(as.ts(observado), name="observado") %>%
hc_add_series(as.ts(previsto), name="previsto") %>%
hc_colors(plotly_palette[c(1,4)]) %>%
hc_title(text = "NOAA - Previsto vs Observado (últimas 12 obs) - Arima(4,0,1)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))X.0 <- X.NASA_ts
N <- length(X.0)
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
fit <- arima(X, order=c(3, 1, 2),xreg=1:n)
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
hchart(as.ts(observado), name="observado") %>%
hc_add_series(as.ts(previsto), name="previsto") %>%
hc_colors(plotly_palette[c(2,4)]) %>%
hc_title(text = "NASA - Previsto vs Observado (últimas 12 obs) - Arima(3,1,2)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))item ii) Previsões (tendências) para os próximos 12 meses
# ajuste
library(forecast)
# NOAAfit <- arima(X.NOAA_ts, order=c(3,0,1)) # erro ! non-stationary AR part from CSS
NOAAfit <- arima(X.NOAA_ts, order=c(4,0,1), method="ML") # mais lento, porem mais acurado
NASAfit <- arima(X.NASA_ts, order=c(3,1,2), method = "ML")Valores Preditos NOAA:
library(forecast)
NOAAfcast <- forecast(NOAAfit, h=12)
as.data.frame(NOAAfcast, rownames = TRUE)Valores Preditos NASA:
NASAfcast <- forecast(NASAfit, h=12)
as.data.frame(NASAfcast, rownames=TRUE)Plots dos valores preditos:
# help(window)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
NOAAfcast_window <- NOAAfcast # para selecionar janelas de tempo em ts
NOAAfcast_window$x <- window(NOAAfcast$x, 2008, 2017)
hchart(NOAAfcast_window, name="NOAA", type="line") %>%
hc_colors(plotly_palette[c(1,5,4,3,6)]) %>%
hc_title(text = "NOAA - Série Histórica Mensal dos Desvios de Temperatura - Previsão 12 Meses (zoom Jan 2008 - Dez 2017)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))# plot(NOAAfcast_window)
NASAfcast_window <- NASAfcast # para selecionar janelas de tempo
NASAfcast_window$x <- window(NASAfcast$x, 2008, 2017)
hchart(NASAfcast_window, name="NASA", type="line") %>%
hc_colors(plotly_palette[c(2,5,4,3,6)]) %>%
hc_title(text = "NASA - Série Histórica Mensal dos Desvios de Temperatura - Previsão 12 Meses (zoom Jan 2008 - Dez 2017)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))# plot(NASAfcast_window)2) Análise dos volumes mensais evaporados no reservatório Echo - Colorado - EUA, desde 1967.
Contextualização do problema:
O arquivo \(\verb|Echo.csv|\) contém a série histórica dos volumes mensais evaporados (em acre-pé) no reservatório Echo, desde 1967, localizado na região do Colorado, nos Estados Unidos.
item i) Identificar um processo SARIMA para a série temporal, justificando a escolha por meio de critérios técnicos.
Dados:
Após removermos a coluna de índices dos dados diretamente no Excel, temos:
# ----- leitura e carregamento
library(tibbletime)
library(dplyr)
library(ggplot2)
library(readr)
library(lubridate)
library(reshape2)
library(highcharter)
echo.loc <- "/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/Echo_alt.csv"
echo <- read_csv(echo.loc)
# ----- data preparation
# eliminando os NA's
# e formatando Data para tibbletime
echo2 <- echo %>%
na.omit() %>%
dplyr::mutate(Date = lubridate::dmy(Date)) %>% # usando dmy do lubridate
as_tbl_time(index = Date)
echo2Começamos pela plotagem das séries, a fim de proceder a uma análise exploratória.
Gráficos das séries mensais:
# crinado as ts
echo_ts <- ts(echo2 %>% dplyr::select(X), start = c(1967,1), end =c(2018,1), frequency = 12)library(forecast)
library(highcharter)
library(ggplot2)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
hchart(echo_ts) %>%
hc_colors(plotly_palette[5]) %>%
hc_title(text = "Série Histórica Mensal de Evaporação - Reservatório Echo (1967 - 2018)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))Gráfico da série semestral:
# ----- plots serie:
# --- serie:
# ggplot:
p1_smst <- echo2 %>%
as_period("6 months") %>% # haverah uma linha para cada ano
ggplot() +
geom_line(aes(x = Date, y = X), size=0.3, alpha = 0.5, colour=plotly_palette[5]) +
labs(x="Data", y="Valores")+
ggtitle("Série Histórica Semestral\nReservatório Echo (1967 - 2018)")+
theme_bw()+
theme(panel.border = element_blank())+ # para ficar igual o plotly
guides(color=guide_legend(title=NULL))+
# scale_color_manual(values=plotly_palette[4])+
theme(plot.title = element_text(hjust=0.5))
plotly::ggplotly(p1_smst) %>%
plotly::layout(legend = list(orientation = "h", x = 0.37, y =-0.1))Gráfico da série anual:
# ----- plots serie:
# --- serie:
# ggplot:
p1_ano <- echo2 %>%
as_period("1 year") %>% # haverah uma linha para cada ano
ggplot() +
geom_line(aes(x = Date, y = X), size=0.3, alpha = 0.5, colour=plotly_palette[5]) +
labs(x="Data", y="Valores")+
ggtitle("Série Histórica Anual\nReservatório Echo (1967 - 2018)")+
theme_bw()+
theme(panel.border = element_blank())+ # para ficar igual o plotly
guides(color=guide_legend(title=NULL))+
# scale_color_manual(values=plotly_palette[4])+
theme(plot.title = element_text(hjust=0.5))
plotly::ggplotly(p1_ano) %>%
plotly::layout(legend = list(orientation = "h", x = 0.37, y =-0.1))Como não conhecemos muito bem a série, optamos por fazer um gráfico das médias mensais. Isso ajudará a confirmarmos um eventual comportamento sazonal.
Plot das médias mensais:
# ----- plots serie:
# --- serie:
# ggplot:
p1_medias <- echo2 %>%
dplyr::mutate(month = format(Date, "%m"), year = format(Date, "%Y")) %>%
group_by(month) %>%
dplyr::summarise(medias = mean(X)) %>%
ggplot() +
geom_line(aes(x = month, y = medias, group=1), size=0.7, alpha = 0.5, colour=plotly_palette[5]) +
labs(x="Data", y="Valores")+
ggtitle("Médias Mensais\nReservatório Echo (1967 - 2018)")+
theme_bw()+
theme(panel.border = element_blank())+ # para ficar igual o plotly
guides(color=guide_legend(title=NULL))+
# scale_color_manual(values=plotly_palette[4])+
theme(plot.title = element_text(hjust=0.5))
plotly::ggplotly(p1_medias) %>%
plotly::layout(legend = list(orientation = "h", x = 0.37, y =-0.1))
Nos gráficos mensal, semestral e anual já é possível notar a presença de sazonalidade. Dirimindo quaisquer dúvidas sobre esta última constatação, o gráfico das médias mensais mostra que há diferenças entre os meses e, portanto, sazonalidade na série. Neste último gráfico, assim como no mensal e no semestral, verifica-se que a evaporação sobe no primeiro semestre de cada ano, atingindo o pico no mês de julho (período mais quente no hemisfério norte) para em seguida declinar, atingindo o menor valor para o ano no mês de dezembro (período mais frio).
O gráfico anual serve apenas para uma análise mais completa e de modo que pudéssemos eventualmente verificar possíveis ciclos entre os anos. Com exceção do pico no ano de 1986 (que provavelmente não é uma quebra estrutural), a série, em termos anuais, parece ser bem comportada.
A série não parece indicar a presença de qualquer tendência. Para confirmar novamente a existência de sazonalidade e eliminar quaisquer dúvidas acerca de uma eventual tendência, utilizamos os plots da FAC e FACP diferenciada de lag S=12. Este valor deve-se ao fato de que, por termos uma série mensal - que apresentou comportamento sazonal dentro do ano (diferenças entre os meses), espera-se que os valores de evaporação do mesmo mês do ano seguinte estejam correlacionados aos valores do mesmo mês do último ano. Este plot também nos ajudará a identificar componentes tanto não-sazonais quanto sazonais de nosso modelo ARIMA(p,i,q)\(\times\)(P,D,Q)\(_{[12]}\).
Plots da FAC e FACP diferenciada para S=12:
library(gridExtra)
grid.arrange(gg_acf(diff(echo_ts, lag=12), title = "FAC - Echo", lag.max=12*5), gg_pacf(diff(echo_ts, lag=12), title = "FACP - Echo", lag.max=12*5), ncol=2)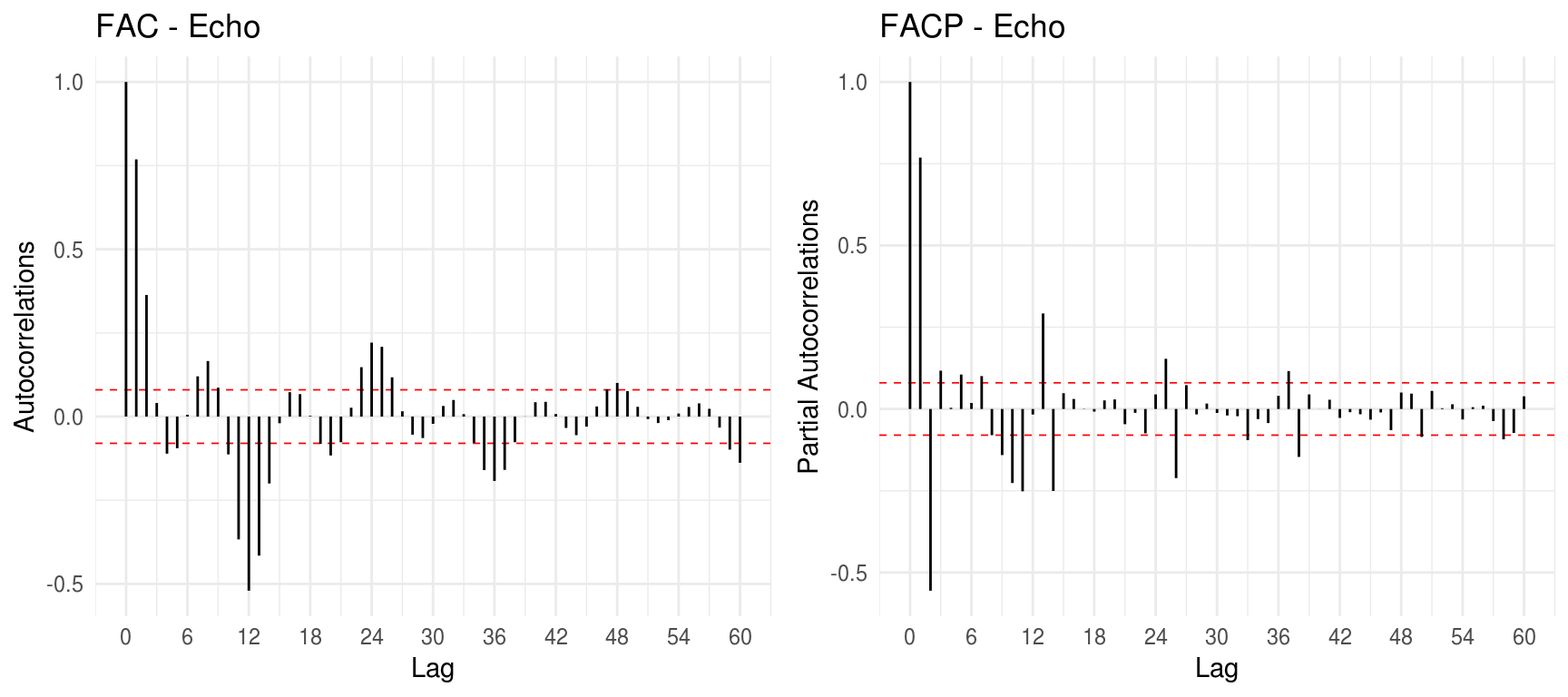
Na FACP e na FAC, na parte não-sazonal, há picos mais significantes nos lags 1, 2 e 11 assim como na FAC. Já na porção sazonal da FAC, temos picos claros que vão reduzindo de intensidade em múltiplos de \(S=12\).
Passamos, então, ao ajuste dos Modelos SARIMA para nossos dados.
Ajuste do modelo:
Algoritmo para testar várias combinações de modelo até ordem 3 e calcular o BIC:
library(astsa)
# ordens que queremos testar
x <- rep(0:3, 2)
# ou # expand.grid(df = data.frame(x=0:3, y=0:3))
cb1 <- combn(x, 2)
cb1 <- t(unique(t(cb1)))
cb1 <- cb1[,-3] # eliminando coluna soh de zeros
# cb1 <- cb1[,-c(3,4,5,9,13,14,15)]
# cb2 <- combn(rev(x), 2)
# cb1[1,5] <- "a"
cb2 <- cb1
BIC.v <- NULL
k = 0
model.orders <- NULL
# library(astsa)
for(j in 1:ncol(cb1)){
for(J in 1:ncol(cb2)){
p <- cb1[1,j]
q <- cb1[2,j]
P <- cb2[1,J]
Q <- cb2[2,J]
k <- k + 1
model.orders[k] <- paste(p,0,q,P,1,Q, sep=",")
# assign(paste0("m.", k), sarima(echo_ts, p,0,q, P,1,Q, S=12, details=FALSE))
BIC.v[k] <- tryCatch({
m <- sarima(echo_ts, p,0,q, P,1,Q, S=12, details=FALSE)
m$BIC
},
error = function(e){
"erro"
})
}
}
dfBIC <- data.frame(model = model.orders, BIC = BIC.v)
# ordenando
dfBIC2 <- dfBIC %>%
dplyr::mutate(BIC = as.numeric(as.character(BIC))) %>%
dplyr::arrange(BIC, model )
readr::write_tsv(dfBIC2, path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/dfBIC2.txt")Modelos testados em ordem de menor BIC:
dfBIC2 <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/dfBIC2.txt") %>%
as.data.frame() %>%
dplyr::mutate(BIC = round(BIC, 4))
dfBIC2Passamos agora ao cálculo dos MAPE dos modelos adotados como possíveis candidatos no cálculo do BIC.
library(astsa)
# X <- as.ts(echo$X)
# tanto faz
X <- echo2$X
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
# fit <- arima(X, order=c(2,0,3), seasonal=list(order=c(3,1,2), period=12),
# method="CSS-ML")
# fit2 <- sarima(X, 2,0,3,3,1,2, S=12)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
2,0,3,3,1,2, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
# mape.203312 <- mean(abs.pred.error)
mape.203312 <- "erro optim"
# ------------------------
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
3,0,2,3,1,2, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
mape.302312 <- mean(abs.pred.error)
# ------------------------
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
2,0,3,3,1,3, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
mape.203313 <- mean(abs.pred.error)
# ------------------------
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
3,0,3,3,1,2, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
mape.303312 <- mean(abs.pred.error)
mape.303312 <- "erro optim"
# ------------------------
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
3,0,0,3,1,3, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
mape.300313 <- mean(abs.pred.error)
# ------------------------
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
3,0,0,2,1,3, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
mape.300213 <- mean(abs.pred.error)
mape.sarima <- data.frame(mape = c(mape.203312, round(mape.302312,4), round(mape.203313,4), mape.303312, round(mape.300313,4), round(mape.300213,4)))
readr::write_tsv(mape.sarima, path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/mape_sarima.txt")Grid com o melhor modelo:
mape.sarima <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/mape_sarima.txt")
# nao eh numeric
df.result <- cbind(dfBIC2[1:6,], mape.sarima)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
library(knitr)
library(kableExtra)
df.result %>%
dplyr::mutate(
mape = cell_spec(mape,
background = ifelse(mape == "0.1744", plotly_palette[3], "white"),
color = dplyr::if_else(mape == "0.1744", "white", "black"))) %>%
# mape nesse caso eh string, nao eh numero
kable(format="html", escape=F, digits = 4, caption="Grid SARIMA") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = T) #%>%| model | BIC | mape |
|---|---|---|
| 2,0,3,3,1,2 | 5.4907 | erro optim |
| 3,0,2,3,1,2 | 5.4937 | 0.1752 |
| 2,0,3,3,1,3 | 5.4979 | 0.1744 |
| 3,0,3,3,1,2 | 5.5014 | erro optim |
| 3,0,0,3,1,3 | 5.5110 | 0.1803 |
| 3,0,0,2,1,3 | 5.5162 | 0.1826 |
Portanto, o modelo mais adequado ao caso, utilizando como critério o MAPE seria o ARIMA(2,0,3)\(\times\)(3,1,3)\(_{[12]}\).
Plot valores preditos vs. observados nos modelos selecionados:
Abaixo apresentamos os plots comparando os valores observados e valores preditos para as últimas 12 observações para o modelo selecionado.
library(astsa)
X <- echo2$X
abs.pred.error <- NULL
pred <- NULL
n <- length(X)
for(t in 0:11){
model <- sarima.for(X[1:(n-12+t)], n.ahead=1,
2,0,3,3,1,3, S=12, newxreg = n+1)
abs.pred.error[t+1] <- abs((X[(n-12+t+1)]-as.numeric(model$pred))/X[(n-12+t+1)])
pred[t+1] <- as.numeric(model$pred)
}
X.last.12 <- X[(n-11):n]
obs_pred.sarima <- data.frame(X.last.12, pred)
readr::write_tsv(obs_pred.sarima, path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/obs_pred.sarima.txt")obs_pred.sarima <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/obs_pred.sarima.txt")
X.last.12 <- obs_pred.sarima$X.last.12
pred <- obs_pred.sarima$predhchart(as.ts(X.last.12), name="observado") %>%
hc_add_series(as.ts(pred), name="previsto") %>%
hc_colors(plotly_palette[c(5,4)]) %>%
hc_title(text = "Echo - Previsto vs Observado (últimas 12 obs) - Arima(2,0,3)x(3,1,3)[12]",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))item ii) Previsões (perfil sazonal) para os próximos 12 meses
library(forecast)
library(astsa)
echofit <- arima(echo_ts, order=c(2,0,3), seasonal=list(order=c(3,1,3),period=12)) # usaremos arima para facilitar o plot
echofcast <- forecast(echofit, h=12)Valores Preditos:
as.data.frame(echofcast, rownames = TRUE)Plots dos valores preditos:
# help(window)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
echofcast_window <- echofcast # para selecionar janelas de tempo em ts
echofcast_window$x <- window(echofcast$x, 2010, 2018)
hchart(echofcast_window, name="NOAA", type="line") %>%
hc_colors(plotly_palette[c(5,1,4,3,6)]) %>%
hc_title(text = "Série Histórica Mensal de Evaporação - Previsão 12 Meses (zoom Jan 2010 - Dez 2018)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))# plot(echofcast)3) Análise dos níveis máximos anuais do Rio Nilo (622 D.C. - 1284 D.C.)
Contextualização do problema:
O arquivo \(\verb|DadosNilo2.txt|\) contém a série histórica dos níveis máximos anuais observados no Rio Nilo, Egito, do ano 622 D.C. ao ano 1284 D.C.
item i) Identificar um processo ARIMA para essa série temporal, justificando a escolha por meio de critérios técnicos.
Dados brutos antes do tratamento:
# ----- leitura e carregamento
library(tibbletime)
library(dplyr)
library(ggplot2)
library(readr)
library(lubridate)
library(reshape2)
nilo.loc <- "/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/DadosNilo2.txt"
nilo <- as.data.frame(scan(nilo.loc, what = list(volume=0)))
# nilo[is.na(nilo),]
niloNosso interesse é obter o melhor modelo em termos técnicos, ou seja, obter um modelo ARIMA com o melhor desempenho de predição. Por isso, nossa estratégia não será a de testar a estacionariedade da série, mas sim ajustar modelos com a série sem diferenciar e com a série diferenciada e verificar seus resultados em termos do Mean Absolute Percentage Error (MAPE) na predição de um passo.
Inicialmente, procedemos a uma análise de ordem exploratória nos dados para termos uma ideia do comportamento da série.
Começamos pela plotagem das séries.
Gráficos das séries mensais:
# crinado as ts
nilo_ts <- ts(nilo %>% dplyr::select(volume), start = 622, end = 1284, frequency = 1) # uma por anolibrary(forecast)
library(highcharter)
library(ggplot2)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
hchart(nilo_ts, nome="volume") %>%
hc_colors(plotly_palette[6]) %>%
hc_title(text = "Série Histórica Nível Máximo Anual do Rio Nilo (622 D.C. - 1284 D.C.)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))Plots da FAC e FACP:
library(gridExtra)
grid.arrange(gg_acf(nilo_ts, title = "FAC - Nilo", lag.max=12*10), gg_pacf(nilo_ts, title = "FACP - Nilo", lag.max=12*10), ncol=2)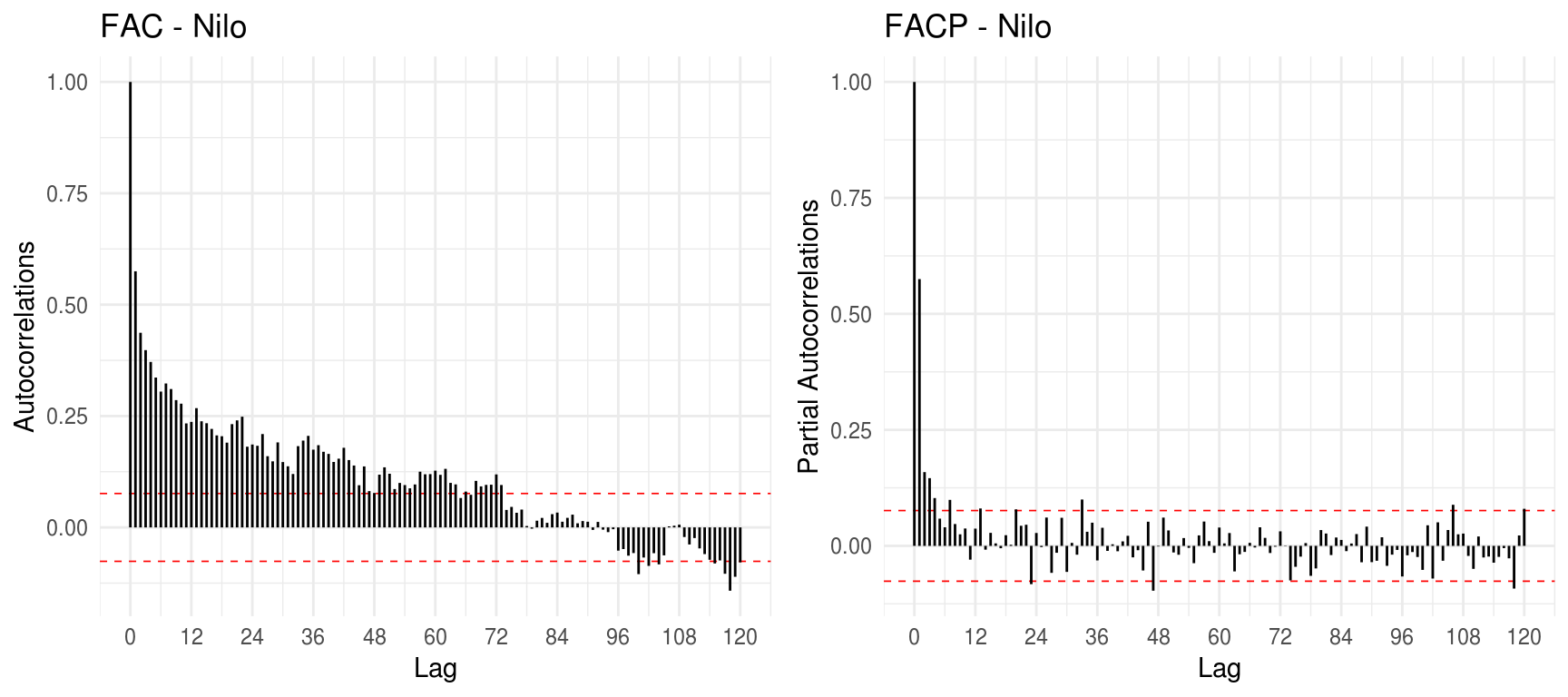
Olhando para o gráfico da série, não parece haver qualquer tendência nos dados. Não há evidências também de sazonalidade, indicando a adequabilidade de um modelo ARIMA não-sazonal para o caso. A FAC apresenta um decaimento relativamente lento, mas que indica provavelmente um modelo autorregressivo de memória longa e talvez não uma ausência de estacionariedade. Como nosso objetivo é obter o modelo com base na abordagem empírica de performance na predição, deixaremos que eventuais restrições relacionadas a uma possível não-estacionariedade da série reflitam nos resultados ao selecionarmos os melhores modelos. Utilizaremos o Mean Absolute Percentage Error (MAPE) de um passo a frente como critério de otimalidade, tomando como base os últimos 12 meses, para confeccionar um grid que nos apontará os modelos candidatos.
Modelos sem diferenciação [ARIMA(p,0,q)]
# -----------------------------------------------------------------------
# Identificao da ordem do processo
# com base no erro de previsao 1 passo a frente
# relativo as ultimas 12 observacoes
# -----------------------------------------------------------------------
X.0 <- nilo$volume # extrair somente os valores como vetor mesmo
N <- length(nilo$volume)
p.max = 7
q.max = 3
MAPE <- matrix(0, p.max+1, q.max+1)
for (p in 0:p.max){
for (q in 0:q.max){
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
# fit <- arima(X, order=c(p, 1, q),xreg=1:n) #Certo
fit <- arima(X, order=c(p, 0, q),xreg=1:n) #Certo
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
MAPE[(p+1),(q+1)] <- 100*(mean(abs((previsto-observado)/observado)))
}
}
MAPE_nodiff_nilo <- MAPE
readr::write_tsv(as.data.frame(MAPE_nodiff_nilo), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_nilo.txt")Temos os seguintes grids resultantes do algoritmo de otimização:
MAPE_nodiff_nilo <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_nodiff_nilo.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(., 4) %>%
`rownames<-`(paste0("p=", 0:7))Para cada série analisada, elencamos os 3 melhores modelo, os quais são destacados nos respectivos grids.
library(kableExtra)
library(knitr)
MAPE_nodiff_nilo %>%
dplyr::mutate(
`q=0` = cell_spec(`q=0`,
background = ifelse(`q=0` == 4.3837, plotly_palette[3],
ifelse(`q=0` < 4.45 & `q=0` != 4.3837, plotly_palette[6], "white")),
color = dplyr::if_else(`q=0` < 4.45, "white", "black")),
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` < 4.5, plotly_palette[6], "white"),
color = dplyr::if_else(`q=1` < 4.5, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid Nilo - série não diferenciada") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 5.6908 | 4.7865 | 4.9811 | 4.7038 |
| p=1 | 4.7187 | 4.4472 | 4.6277 | 4.6964 |
| p=2 | 4.6182 | 4.712 | 4.7140 | 4.6784 |
| p=3 | 4.3837 | 4.7184 | 4.7156 | 4.7001 |
| p=4 | 4.4488 | 4.6782 | 4.6954 | 4.8988 |
| p=5 | 4.4835 | 4.5002 | 4.6670 | 4.8862 |
| p=6 | 4.4702 | 4.683 | 4.6671 | 4.8381 |
| p=7 | 4.6226 | 4.744 | 4.7561 | 4.8528 |
Após selecionarmos os melhores modelos no caso da série não diferenciada, passamos para a confecção dos grids de desempenho para a série diferenciada.
Modelos com diferenciação de ordem 1 [ARIMA(p,1,q)]
Novamente, utilizaremos o Mean Absolute Percentage Error (MAPE) de um passo a frente como critério de otimalidade e tomando como base os últimos 12 meses para confeccionar um grid que nos apontará os modelos candidatos.
# -----------------------------------------------------------------------
# Identificao da ordem do processo
# com base no erro de previsao 1 passo a frente
# relativo as ultimas 12 observacoes
# -----------------------------------------------------------------------
X.0 <- nilo$volume # extrair somente os valores como vetor mesmo
N <- length(nilo$volume)
p.max = 7
q.max = 3
MAPE <- matrix(0, p.max+1, q.max+1)
for (p in 0:p.max){
for (q in 0:q.max){
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
fit <- arima(X, order=c(p, 1, q),xreg=1:n) #Certo
# fit <- arima(X, order=c(p, 0, q),xreg=1:n) #Certo
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
MAPE[(p+1),(q+1)] <- 100*(mean(abs((previsto-observado)/observado)))
}
}
MAPE_diff_nilo <- MAPE
readr::write_tsv(as.data.frame(MAPE_diff_nilo), path="/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_nilo.txt")Temos os seguintes grids resultantes do algoritmo de otimização:
MAPE_diff_nilo <- readr::read_tsv("/home/allan/Documents/1S2018/A_SERIES_TEMPORAIS/PP2/dados/MAPE_diff_nilo.txt") %>%
magrittr::set_colnames(paste0("q=", 0:3)) %>%
as.data.frame() %>%
round(., 4) %>%
`rownames<-`(paste0("p=", 0:7))Para cada série analisada, elencamos os 3 melhores modelo, os quais são destacados nos respectivos grids.
library(kableExtra)
library(knitr)
MAPE_diff_nilo %>%
dplyr::mutate(
`q=0` = cell_spec(`q=0`,
background = ifelse(`q=0` == 4.0483, plotly_palette[3],
ifelse(`q=0` < 4.45 & `q=0` != 4.3837, plotly_palette[6], "white")),
color = dplyr::if_else(`q=0` < 4.45, "white", "black")),
`q=1` = cell_spec(`q=1`,
background = ifelse(`q=1` < 4.5, plotly_palette[6], "white"),
color = dplyr::if_else(`q=1` < 4.5, "white", "black")),
ordem = rownames(.),
ordem = cell_spec(ordem, color = "black")) %>%
dplyr::select(ordem, paste0("q=", 0:3)) %>%
kable(format="html", escape=F, digits = 4, caption="grid Nilo - série diferenciada") %>%
kable_styling(bootstrap_options = c("striped", "hover"), full_width = F) #%>%| ordem | q=0 | q=1 | q=2 | q=3 |
|---|---|---|---|---|
| p=0 | 4.9889 | 4.745 | 4.8405 | 4.9001 |
| p=1 | 4.7124 | 4.8692 | 4.8346 | 4.7068 |
| p=2 | 4.0483 | 4.8607 | 4.8579 | 4.7071 |
| p=3 | 4.3445 | 4.7404 | 4.7331 | 4.7660 |
| p=4 | 4.3819 | 4.7299 | 4.7318 | 5.1384 |
| p=5 | 4.2914 | 4.7308 | 4.7468 | 4.9004 |
| p=6 | 4.5855 | 4.7521 | 4.7778 | 4.9065 |
| p=7 | 4.6389 | 4.7937 | 4.8062 | 4.9051 |
Dentre os modelos analisados (para série não diferenciada e diferenciada), o melhor modelo, utilizando como critério o desempenho medido pelo MAPE de um passo é um ARIMA(2,1,0), o que se aproxima do modelo sugerido por Aguado (1982), qual seja, Arima(2,1,1). A diferença deve-se provavelmente ao fato de Aguado (1982) ter utilizado como critério o ajuste do modelo comporando os BIC’s, enquanto nós utilizamos o MAPE, focando no desempenho preditivo.
Plot valores preditos vs. observados nos modelos selecionados:
Abaixo apresentamos os plots comparando os valores observados e valores preditos para as últimas 12 observações para o modelo selecionado.
X.0 <- nilo_ts
N <- length(X.0)
# X.0 <- nilo$volume # extrair somente os valores como vetor mesmo
# N <- length(nilo$volume)
previsto <- NULL
observado <- NULL
for (t in 0:11){
X <- X.0[1:(N-12+t)]
n <- length(X)
fit <- arima(X, order=c(2, 1, 0),xreg=1:n)
prev <- predict(fit, n.ahead = 1, newxreg=(n+1))
obs <- X.0[N-12+t+1]
previsto[t+1] <- prev$pred
observado[t+1] <- obs
}
hchart(as.ts(observado), name="observado") %>%
hc_add_series(as.ts(previsto), name="previsto") %>%
hc_colors(plotly_palette[c(6,4)]) %>%
hc_title(text = "Nível Máximo Nilo - Previsto vs Observado (últimas 12 obs) - Arima(2,1,0)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))item ii) Previsões para os próximos 5 anos
library(forecast)
library(astsa)
nilofit <- arima(nilo_ts, order=c(2,1,0)) # usaremos arima para facilitar o plot
nilofcast <- forecast(nilofit, h=5)Valores Preditos:
as.data.frame(nilofcast, rownames = TRUE)Plot dos valores preditos:
# help(window)
plotly_palette <- c('#1F77B4', '#FF7F0E', '#2CA02C', '#D62728', "#9467bd", "#8c564b")
nilofcast_window <- nilofcast # para selecionar janelas de tempo em ts
nilofcast_window$x <- window(nilofcast$x, 1180, 1289)
hchart(nilofcast_window, name="NOAA", type="line") %>%
hc_colors(plotly_palette[c(6,1,4,3,5)]) %>%
hc_title(text = "Série Histórica Anual - Nível Rio Nilo - Previsão 5 Anos (zoom 1180 - 1289)",
margin = 20, align = "center",
style = list(color = "black", useHTML = TRUE))# plot(echofcast)Referências Bibliográficas
AGUADO, E. Time Series Analysis of Nile Flows. Annals of the Association of the American Geographers 72: 109-19.
MORETTIN, P.A. & TOLOI, C.M. Análise de Séries Temporais. 2a ed., Edgard Blücher, 2005.
NOTAS DE AULA. Análise de Séries Temporais. Curso de Graduação em Estatística, UnB, 2018.
R CORE TEAM. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL https://www.R-project.org/.
SHUMWAY, R.H. & STOFFER, D.S. Time Series Analysis and Its Applications with R Examples. Springer, 2011.